https://www.riken.jp/press/2024/20240219_3/index.html
https://www.riken.jp/press/2024/20240219_3/index.html
理化学研究所(理研)生命医科学研究センター 統合細胞システム研究チームの柚木 克之 チームリーダーらの研究チームは、細胞内の生体分子の大規模な相互作用ネットワークである多階層ネットワーク[1]を、人間が解釈しやすい形で自動的に可視化するソフトウエア「transomics2cytoscape」を開発・公開しました。
本研究成果は、データ駆動型生命医科学の確立に向けた大規模データの統合解析および可視化、知識発見に貢献すると期待されます。
DNAやタンパク質の測定技術の発展に伴い、生命医科学分野においては、細胞や組織で発現する分子を網羅的に計測するオミクス[1]研究が広く行われています。オミクス研究により、生体分子の相互作用ネットワークを調べることができますが、ネットワークの可視化技術は属人的な手作業に依存しており、多大な時間と労力がかかるという課題がありました。
今回、研究チームは、過去に発表されたトランスオミクス[1]研究のデータセットからその可視化のワークフローを一般化・自動化し、オープンなソフトウエアとして無償公開しました。このソフトウエアにより、これまでは多大な負担と時間を要し、再現も困難だった多階層ネットワークの可視化を全自動で実施できます。
本研究は、科学雑誌『npj Systems Biology and Applications』オンライン版(2月19日付:日本時間2月19日)に掲載されました。
背景
細胞や組織で発現する分子を網羅的に計測するオミクス解析は、生命機能の全体像(「森」)を把握し、個々の分子や遺伝子(「木」)が動作する文脈を理解する、いわば「森と木をつなぐ」上で重要です。生体分子の相互作用ネットワークの可視化はオミクスデータの解釈や理解を多人数で共有するための中心的技術です。柚木チームリーダーらは2014年の論文注1)で、リン酸化プロテオーム[2]およびメタボローム[2]をトランスオミクスの方法論に則って統合し、インスリンが複数のオミクス階層にまたがって作用するメカニズムを示すネットワーク(多階層ネットワーク)を再構築・可視化しました。しかし、この多階層ネットワークの可視化は属人的なノウハウに依存しており、多大な時間と労力を要するという難点がありました。本研究では近年のワークフロー自動化の潮流を踏まえ、属人的な手作業を脱するためのソフトウエア「transomics2cytoscape」の開発を行いました。
- 注1)Yugi, K., Kubota, H., Toyoshima, Y., Noguchi, R., Kawata, K., Komori, Y., Uda, S., Kunida, K., Tomizawa, Y., Funato, Y., Miki, H., Matsumoto, M., Nakayama, K.I., Kashikura, K., Endo, K., Ikeda, K., Soga, T., Kuroda, S., "Reconstruction of insulin signal flow from phosphoproteome and metabolome data", Cell Rep. 8(4), 1171-1183, 2014.
研究手法と成果
多階層ネットワークの可視化にあたり、研究者が利用しやすい回路図を描くため、研究者が元々持っているイメージ(事前知識)の活用を原則としました。活用した事前知識は「生化学パスウェイ[3]の慣習的なレイアウト」と「オミクス階層図」です。
生化学パスウェイの慣習的なレイアウトとは、例えば代謝に関わる研究者に共有されている代謝経路の大まかなマップを指します。身近な図で例えると、大都市の地下鉄利用者は、慣習的な路線図のレイアウトを事前知識として持っており、目的地までの乗り換えに役立てています(図1a)。しかし、従来のネットワーク可視化ソフトウエアで自動的に路線図を描かせると、何らかのアルゴリズムに従って路線図のレイアウトを決めてしまうことが大半であるため、慣習的な路線図とはかけ離れたものになってしまいます(図1b、c)。
生化学パスウェイも事情は地下鉄と同様です。代謝マップ(図1d)があれば、代謝研究者がまず探すのは慣習的に円として描かれるTCAサイクル(クエン酸回路)[4]の位置です。これを基準として、TCAサイクルから上方に伸びている直線状の経路があると、それが解糖系であろうという当たりを付けます。しかし、従来のソフトウエアは何らかのアルゴリズムに従ってネットワークのレイアウトを再配置してしまうため、慣習的なレイアウトに則ってデータを解釈することが困難でした(図1e、f)。
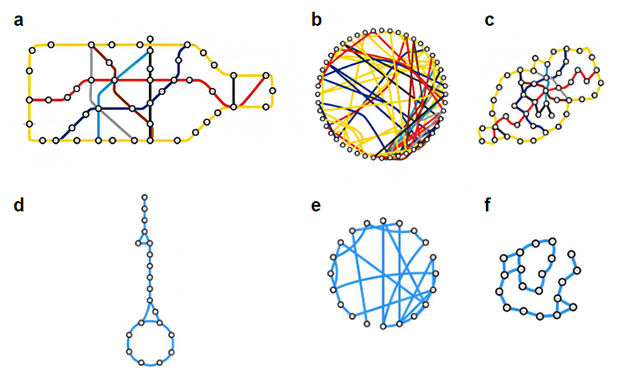
図1 レイアウトの「表現」によってイメージが変わる地下鉄路線図(上)と代謝マップ(下)
aはロンドンの地下鉄路線図。bとcはサークルレイアウトとCompound Spring Embedder(CoSE)レイアウトでそれぞれ表現した地下鉄路線図。利用客はオリジナルの路線図であるaの配置に慣れている。しかし、自動レイアウトアルゴリズム(b、c)ではそれとは似ても似つかないレイアウトになる。dは基本的な生化学パスウェイである解糖系とTCAサイクルの慣習的なネットワークレイアウト。eとfはサークルレイアウトとCoSEレイアウトを解糖系とTCAサイクルに対して適用したものである。
一方、オミクス階層図は、ゲノム[2]、トランスクリプトーム[2]、プロテオーム、メタボロームなどの生体分子の分類もしくは機能分類を各層ごとに表現し、遺伝情報の伝達順序に沿って層状に重ねた図を指し、いわゆる統合オミクス解析の概念図として用いられています。
本研究では生化学パスウェイの慣習的なレイアウトを保ちつつオミクス階層図を作り、オミクス階層間の相互作用(トランスオミクス相互作用)を自動的に可視化するソフトウエア「transomics2cytoscape」を開発しました。transomics2cytoscapeにより、既存の可視化ソフトウエアでは不可能だった、人間の事前知識を生かした多階層ネットワークの可視化が実現しました。transomics2cytoscapeの具体的な利用例として、これまでに報告されたトランスオミクス研究における多階層パスウェイの手作業での可視化を、手作業無しで自動的に再現可能であることを示しました(図2)。
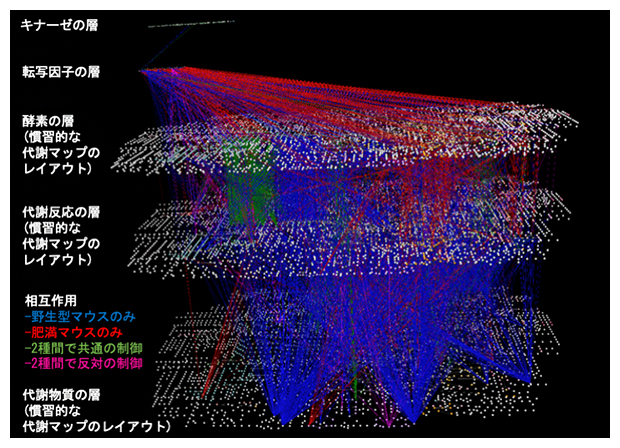
図2 糖尿病マウスと野生型マウスにおけるキナーゼの多階層ネットワーク
transomics2cytoscapeによって、これまでに報告されていたトランスオミクス研究を踏まえた多階層ネットワークを可視化した。これは従来手作業で構築されていた多階層ネットワークをよく再現している。
transomics2cytoscapeはオープンソースソフトウエアの開発を目的としたプロジェクト「Bioconductor[5]」の1パッケージとして登録されているため、再現性や定期的な更新に優れ、だれでも利用可能です注2)。
今後の期待
オミクス研究で産出されるデータは、拡大を続け、公開・共有されるようになっています。大量のデータを可視化し、新たな解釈を与えることは、膨大なデータの解析から生体分子の思いがけない関係性を見いだし、解釈を深めていくデータ駆動型生命医科学を推し進める上でのキーテクノロジーの一つです。本研究では、その解釈を支援するための多階層ネットワーク可視化を自動的に行うソフトウエアを新たに開発・公開し、手作業の労力を省くことに成功しました。これにより、従来のトランスオミクス研究ではプロジェクトごとに1回が限度であった多階層ネットワークの可視化を反復して行えるようになり、解析中のデータからの知識抽出に活用できると期待されます(図3)。
今後は本研究に関連し得るソフトウエアやデータ基盤との接続・統合をさらに進め、既存データリソースやソフトウエアとの相互運用性の向上、VR(仮想現実)中での可視化など本研究を足がかりとしたソフトウエア工学的な研究を展開し、オミクスデータからの発見のサイクルを加速することを目指します。
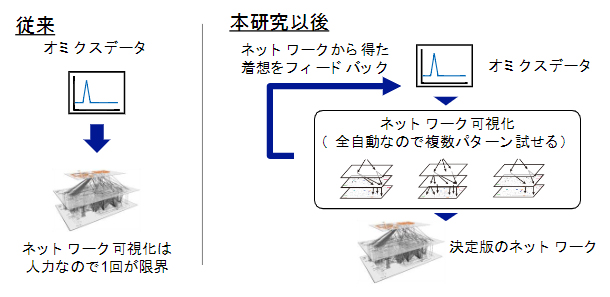
図3 自動化により研究の途中で何度もネットワークの可視化が可能
補足説明
- 1.多階層ネットワーク、オミクス、トランスオミクス
細胞や組織を構成する分子を網羅的に計測して得た情報をオミクスと呼び、DNA、RNA、タンパク質などの異なるオミクス階層を統合的に解析するアプローチをトランスオミクスという。多階層ネットワークとは、異なるオミクス階層にまたがる生化学経路によって特定の生命機能が実現されている仕組みを示すもの。 - 2.リン酸化プロテオーム、メタボローム、ゲノム、トランスクリプトーム
任意の細胞や組織で発現する全リン酸化タンパク質、代謝物、DNA、RNAを指すオミクス。 - 3.パスウェイ
細胞内での化学反応の一連のつながりや、生体分子間の相互作用ネットワークを指す。代謝パスウェイ、シグナル伝達パスウェイ、生体分子の発現の調節パスウェイのようにさまざまなパスウェイが存在し、その理解が細胞の機能や疾患のメカニズムを解明する鍵となり得る。 - 4.TCAサイクル(クエン酸回路)
多くの生物がエネルギーの産出や細胞の原材料を合成するために持つ中心的な代謝パスウェイの一つ。 - 5.Bioconductor
バイオインフォマティクスのためのオープンソースソフトウエアのコレクションかつ世界的なコミュニティ。新しい手法やアルゴリズムが開発されると、それらがBioconductorプロジェクトに追加される。ユーザーと開発者はフォーラムを通じて交流し、ソフトウエアの使用方法や開発についてのサポートを受けることができる。
研究チーム
理化学研究所
生命医科学研究センター 統合細胞システム研究チーム
チームリーダー 柚木 克之(ユギ・カツユキ)
研究パートタイマーⅠ 西田 孝三(ニシダ・コウゾウ)
研究員 丸山 順一(マルヤマ・ジュンイチ)
生命機能科学研究センター バイオコンピューティング研究チーム
上級研究員 海津 一成(カイズ・カズナリ)
チームリーダー 高橋 恒一(タカハシ・コウイチ)
研究支援
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業(CREST)「バイオDX」領域「データ駆動進化医学で解く季節性うつと冬眠の代謝基盤(研究代表者:柚木克之)」、日本学術振興会(JSPS)科学研究費助成事業新学術領域研究(研究領域提案型)「マルチスケール精神病態の構成的理解」における計画研究課題「精神病態の分子基盤解明を可能にする次世代トランスオミクス技術の開発(研究代表者:柚木克之)」、稲盛財団研究助成などによる支援を受けて行われました。
原論文情報
- Kozo Nishida, Junichi Maruyama, Kazunari Kaizu, Koichi Takahashi, Katsuyuki Yugi, "transomics2cytoscape: An automated software for interpretable 2.5-dimensional visualization of trans-omic networks", npj Systems Biology and Applications, 10.1038/s41540-024-00342-8

発表者
理化学研究所
生命医科学研究センター 統合細胞システム研究チーム
チームリーダー 柚木 克之(ユギ・カツユキ)
 柚木 克之
柚木 克之報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム![]()
0 件のコメント:
コメントを投稿