https://www.a.u-tokyo.ac.jp/topics/topics_20250321-1.html
https://www.a.u-tokyo.ac.jp/topics/topics_20250321-1.html
――
発表のポイント
- 遺伝子発現の環境条件に応じた制御に、翻訳抑制・mRNA分解・転写抑制の3つが連動した多層的な制御機構が存在することを発見しました。
- これまで不要と考えられていたmRNA分解中間体からsmall RNA(sRNA)が生成され、NIP5;1遺伝子の転写の抑制に関与していることを発見しました。
- このsRNAは、遺伝子発現を制御するタンパク質ARGONAUTE1(AGO1)に取り込まれ、転写装置であるRNAポリメラーゼII(Pol II)の動きを抑えることで、NIP5;1遺伝子の転写を調節していることを示しました。
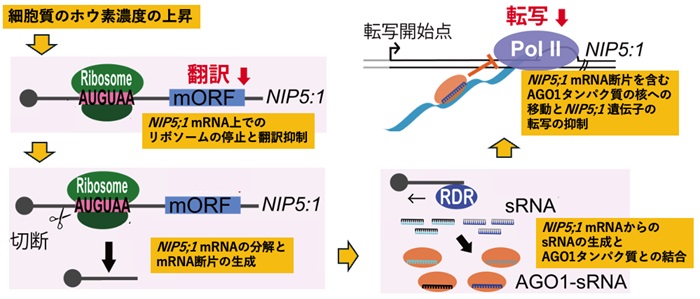
環境中のホウ素濃度が高くなってから、NIP5;1遺伝子の転写の抑制が起こるまでの多層制御機構
研究概要
東京大学大学院農学生命科学研究科の藤原徹教授らは、植物の必須元素の環境中の濃度に応じて適切に吸収を制御するために不可欠な新たな遺伝子発現制御機構を発見しました。具体的にはシロイヌナズナのホウ素輸送体NIP5;1の遺伝子発現が細胞内のホウ素濃度に応じて、転写・翻訳・mRNA分解が連携する「多層的な制御システム」によって調整されていることを今回明らかにしました。特に、5'-非翻訳領域(5'-UTR)(注1)に存在するAUGUAA配列上で、リボソーム(注2)がホウ素濃度を感知して停止することで、翻訳の抑制・mRNAの分解・転写の抑制が連動する仕組みを明らかにしました。
さらに、NIP5;1mRNAの分解が単なる遺伝情報の消去ではなく、転写を調節するシグナルを生み出す役割を持つことを明らかにしました。分解されたmRNAの一部からsRNA(注3)が生成され、NIP5;1の転写を抑制するフィードバック機構として機能していたのです。
本研究により、転写・翻訳・mRNA分解の2つの制御が密接に連携しながら働いていることが示され、これまで個別に研究されることが多かった遺伝子発現の制御が統合的に機能することで環境条件の変化に応じてより効果的に遺伝子の発現を制御していることを明らかにしました。
研究内容
<背景>
ホウ素は植物にとって細胞壁の構造維持に欠かせない栄養素ですが、その量が多すぎても少なすぎても生育に悪影響を及ぼします。そのため、植物は環境の変化に応じてホウ素の吸収を調節する必要があります。しかし、どのようにしてホウ素の量を感知し、それに応じて遺伝子の働きを調整しているのかは、まだ十分に解明されていませんでした。本研究では、モデル植物であるシロイヌナズナがホウ素を土壌から取り込むために必要な輸送タンパク質NIP5;1をコードする遺伝子の発現を調節する新たな遺伝子制御メカニズムを明らかにしました。
<研究内容>
AUGUAA配列を起点とした連携制御
これまでの研究で、NIP5;1の発現量はホウ素濃度に応じて変化し、その調節にはリボソームの停止による翻訳抑制とmRNAの分解が関与していることが分かっていました(Tanaka et al., 2016)。通常、翻訳装置であるリボソームはmRNAの情報を読み取りながらタンパク質を合成します。しかし、NIP5;1遺伝子の5'-UTRにはAUGUAAという開始コドン(注4)と終始コドン(注5)が連続した特徴的な配列が含まれており、細胞内のホウ素濃度が高い場合、この配列上でリボソームが停止し、翻訳が抑制されます。このリボソームの停滞が引き金となり、mRNAの切断、そして分解が促進されることが分かっていました。
本研究では、mRNAの分解中間体からsRNAが生成され、それにより転写の抑制が引き起こされることが明らかになりました。AUGUAA配列を起点として、以下の3段階の統合的な制御が機能することが分かりました。
1. 翻訳の抑制とmRNAの分解
リボソームがAUGUAA上でホウ素濃度依存的に停滞すると、翻訳が抑制されると同時に、AUGUAAの13〜14塩基上流でmRNAの切断が起こり、分解が進行します(Tanaka et al., 2016)。
2. sRNAの生成と転写抑制シグナル
mRNAが分解されると、その一部からsRNAが合成され(下記にて詳しく説明)、NIP5;1遺伝子の転写を抑制するシグナルとして機能することがわかりました。
3. sRNAとAGO1(注6)の結合による転写の抑制
生成されたsRNAは、遺伝子発現の調節を担うタンパク質AGO1と結びつき、転写装置であるRNAポリメラーゼII(注7)の進行を阻害することで、NIP5;1遺伝子の転写を抑制することが分かりました(図1)。
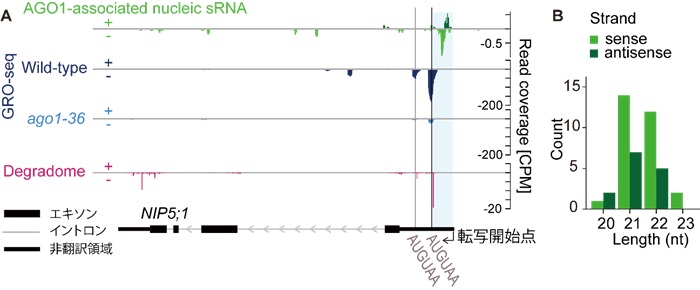
図1:NIP5;1領域において、AGO1と結合したsRNAの蓄積領域(AGO1-associated nucleic sRNA)、RNA polymerase II(Pol II)の活性位置(GRO-seq)、およびmRNA分解中間体の位置(Degradome)を示す。
(A)Degradome解析では、網羅的にmRNA分解中間体の5'末端を検出する。NIP5;1では、AUGUAAの上流でmRNAの切断断片が確認される。また、AGO1-associated nucleic sRNAシークエンス解析は、AGO1タンパク質に結合するsRNAを特異的に精製し、それらの配列を次世代シーケンサーで解析する手法であり、NIP5;1の配列と一致する21〜22塩基のsRNAが転写開始点から1番目のAUGUAA配列までの領域(水色で覆われている領域)で認められる。これらのsRNAはセンス鎖およびアンチセンス鎖の両方に存在したことから、切断領域から二本鎖RNAが合成された可能性が示唆される。GRO-seq解析では、転写が活発に行われている領域を検出する手法であり、Pol IIが存在する位置を明らかにする。今回の結果では、AUGUAA配列周辺にPol IIの顕著な蓄積が観察されたことから、Pol IIの進行がこの領域で停滞していることが示唆される。さらに、AGO1変異株(ago1-36)ではこの蓄積が、野生型と比較して減少している。このことから、AGO1変異株では、AUGUAA上を通過しPol IIが進行していることが推察される。(B) mRNA切断部位の上流で見つかったセンス及びアンチセンスのsRNAの長さの分布を示している。
mRNA分解中間体が遺伝子制御の鍵に!新たな役割の発見
これまで、mRNAの分解中間体は不要なものとして単に排除されると考えられていました。しかし、本研究では、切断された後のmRNA分解中間体の一部からsRNAが合成され、それ自体が転写を抑制するシグナルとして機能することが明らかになりました。
具体的には、細胞質でAUGUAA上にリボソームがホウ素濃度依存的に停滞すると、AUGUAAから13~14塩基上流でmRNAが切断されます。その切断されたmRNAの5'断片(切断された位置から転写開始点(注8)までの領域)が鋳型となり、RNA依存的RNAポリメラーゼ(RDR)(注9)によって二本鎖RNAが合成されます。その後、sRNAを生成する酵素であるDICER-LIKE(DCL)(注10)を介して、21~22 塩基に整えられ、sRNAが生成されていると考えられます。このsRNAはAGO1と結合し、核へと輸送され、sRNAと相補の配列を持つ新生RNAかDNAと結合し、転写装置であるRNA polymerase II(Pol II)の進行を止めることで転写を抑制することが分かりました(図1,図2)。つまり、mRNAの分解は単なる情報の消去ではなく、遺伝子発現を制御するメカニズムの一部に生まれ変わることが示されました。
遺伝子発現制御の新たな視点:統合的な調節メカニズム
近年、転写・翻訳・mRNA分解は、それぞれ独立した制御プロセスであるだけでなく、相互に連携して調節されていることを示唆する報告が増えつつあります。例えば、翻訳と同時にmRNAが分解される共翻訳的mRNA分解(co-translational mRNA degradation)が働くことが報告されています。しかし、このような現象の理解はまだ限定的であり、遺伝子発現の多層的な制御メカニズムには未解明の部分が多く残されています。
本研究では、mRNAの分解中間体からシグナル分子(sRNA)が生成され、核内へ移行し、転写の調節に関与することを明らかにしました。このmRNA分解は、AUGUAA配列上でのリボソームのホウ素濃度依存的な停滞を引き金に起こり、翻訳抑制・mRNA分解・転写制御の3つが連動する多層的な制御メカニズムが形成されることが分かりました。このような多層的な遺伝子発現制御機構は、NIP5;1に特有のものではなく、環境変化に適応するための仕組みとして、他の多くの遺伝子や生物にも広く存在すると考えられます。 今後、この分野の研究が進むことで、生命全体に共通する遺伝子制御の本質に迫る新たな研究展開が期待されます。
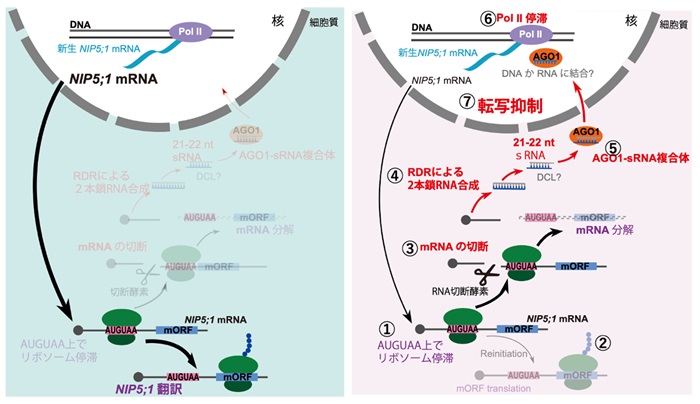
図2 NIP5;1遺伝子発現のホウ素条件に応じた多層的な統合制御機構
左:環境中のホウ素濃度が低い状態でのNIP5;1遺伝子の発現を示す模式図。細胞核に存在するNIP5;1遺伝子からNIP5;1mRNAが合成され、細胞質に移った後に翻訳されてNIP5;1タンパク質が合成される。NIP5;1タンパク質はホウ素の吸収を促進するため細胞のホウ素濃度が適正に保たれる。
右:環境中のホウ素濃度が高くなった時のNIP5;1遺伝子の発現制御を示す模式図。環境中のホウ素濃度が高くなると、細胞質のホウ素濃度も高くなる。細胞質のホウ素濃度が高くなると、NIP5;1mRNAに存在するAUGUAA配列上でリボソームが停滞する(①)。リボソームの停滞により、NIP5;1タンパク質の合成(翻訳)が抑制される(②)。リボソームの停滞はNIP5;1mRNAの切断も引き起こす(③)。切断されたNIP5;1mRNAはRDRの作用で2本鎖RNAとなる(④)。2本鎖RNAとなったNIP5;1mRNA断片は切断を受けた後AGO1タンパク質に取り込まれ、AGO1-sRNA複合体を形成する(⑤)。AGO1-sRNA複合体は核に移行し、Pol IIの進行を停滞させ(⑥)、NIP5;1遺伝子の転写を抑制する(⑦)
図中の赤い矢印は、mRNAの分解断片の一部がAGO1に取り込まれ、最終的に核へと移行する過程を、紫色の部分は、リボソーム停滞とmRNA分解に関する、これまでに報告された研究結果に基づいたプロセスを、本研究で新たに明らかになったプロセスは赤で示している。さらに、灰色で示された「?」となっているプロセスは、今後の研究によって検証が必要な仮説的なメカニズムを示している。また、透明度の高い、左右を比較して薄くなっている部分は、そのプロセスの活性が低いことを示し、矢印の太さは活性の強さを表している。
発表者
東京大学 大学院農学生命科学研究科 応用生命化学専攻
田中 真幸 特任助教:研究当時
現: 大阪公立大学大学院農学研究科 応用生物科学専攻 研究員
藤原 徹 教授
大阪公立大学大学院農学研究科 応用生物科学専攻
反田 直之 助教
北海道大学
大学院農学研究院 応用生命科学分野
尾之内 均 教授
内藤 哲 農学研究院研究員
大学院理学研究院 生物科学部門
千葉 由香子 教授
兼:生命科学研究院 生命システム科学コース 教授
Cardiff University School of Biosciences
Athanasius F. M. Marée Professor
Verônica A. Grieneisen Reader
John Innes Centre Department of Cell and Developmental Biology
Susan Duncan Postdoctoral Scientist
論文情報
- 雑誌
- Nucleic Acids Research (2025)
- 題名
- Ribosome stalling-induced NIP5;1 mRNA decay triggers ARGONAUTE1-dependent transcription down-regulation
- 著者
- Mayuki Tanaka, Naoyuki Sotta*, Susan Duncan, Yukako Chiba, Hitoshi Onouchi, Athanasius F. M. Marée*, Satoshi Naito, Verônica A. Grieneisen*, Toru Fujiwara*,(*責任著者)
- DOI
- 10.1093/nar/gkaf159
- URL
- https://academic.oup.com/nar/article-lookup/doi/10.1093/nar/gkaf159
研究助成
本研究は、科研費「基盤研究(S) 植物の栄養感知機構の解明と栄養応答統御(研究代表者:藤原徹)(課題番号:19H05637)」、「学術変革領域(A)周囲環境応答としての植物成長特性の力学的最適化の柔軟性(研究代表者:藤原徹)(課題番号:18H05490)」、「基盤研究(C) AUG-UAAを介したリボソーム停滞のホウ素栄養制御機構(研究代表者:田中真幸)(課題番号:18K06278)」の支援により実施されました。
用語解説
- 注1) 5'-非翻訳領域(5'-UTR)
mRNAの転写開始点から翻訳開始点(タンパク質の合成が始まる位置)までの間に存在する、タンパク質に翻訳されない領域。この領域は、mRNAの安定性や翻訳効率の調節に関与し、遺伝子発現の制御に重要な役割を果たす。 - 注2) リボソーム
細胞質に存在するタンパク質合成のための装置。mRNAの遺伝情報を読み取り、それに基づいてアミノ酸をつなげ、タンパク質を合成する。 - 注3) small RNA (sRNA)
長さが20~30塩基程度の短いRNA分子で、遺伝子の発現調節に関与する。特定のmRNAと結合することで、その分解を促したり、翻訳を抑制したりする。植物や動物を含む多くの生物で、遺伝子発現の制御に重要な役割を果たしている。 - 注4) 開始コドン
開始コドン(start codon)は、mRNAの翻訳が開始される場所を示す特定の塩基配列。通常、AUGの配列が開始コドンとして機能し、メチオニン(Met)というアミノ酸の合成を指示します。リボソームはこの開始コドンを認識し、mRNAの情報をもとにタンパク質合成を開始します。 - 注5) 終止コドン
終止コドン(stop codon)は、mRNAの翻訳が終了する位置を示す塩基配列です。終止コドンには UAA、UAG、UGA の3種類があり、これらはどのアミノ酸にも対応せず、リボソームが翻訳を終了するシグナルとして機能します。 - 注6) ARGONAUTE1(AGO1)
sRNAと結合し、ターゲットとなるmRNAの分解や翻訳抑制を行うタンパク質。 - 注7) RNAポリメラーゼII(Pol II)
DNAの情報をmRNAへと転写する酵素の一種。真核生物において、タンパク質をコードする遺伝子のmRNAを合成する主要な転写酵素であり、遺伝子発現の中心的な役割を担う。 - 注8) 転写開始点
DNAからmRNAが作られる(転写される)ときに、RNAポリメラーゼが転写を開始する位置。転写開始点より下流の配列がmRNAとして合成され、遺伝子の発現が始まる。 - 注9) RNA依存的RNAポリメラーゼ(RDR)
RNAを鋳型(テンプレート)として新たなRNAを合成する酵素。特に、二本鎖RNA(dsRNA)の合成を行う重要な役割を持つ。 - 注10) DCL(DICER-LIKE)
RNAを特定の長さに切断する酵素で、特sRNAの生成に関与する。 - 注11:図1) イントロン
遺伝子から転写されたRNAのうち、成熟mRNAから除去される部分のこと - 注12:図1) エキソン
遺伝子のDNA配列の中で、タンパク質に翻訳されるコーディング領域(CDS)と、翻訳されない非翻訳領域で構成される。mRNAとして転写された後、不要なイントロンが取り除かれ、エキソンがつなぎ合わさって成熟mRNAとなる。
問い合わせ先
(研究内容については発表者にお問合せください)
東京大学 大学院農学生命科学研究科
応用生命化学専攻 植物栄養・肥料学研究室 教授 藤原 徹(ふじわら とおる)
Fax: 03-5841-8032
大阪公立大学大学院農学研究科
応用生物科学専攻 植物栄養学研究グループ 助教 反田 直之(そった なおゆき)
〈報道に関する問合せ〉
東京大学大学院農学生命科学研究科・農学部
事務部 総務課総務チーム 総務・広報情報担当(広報情報担当)
Tel: 03-5841-8179 FAX: 03-5841-5028
E-mail : koho.a[at]gs.mail.u-tokyo.ac.jp
大阪公立大学 広報課
Tel: 06-6967-1834
E-mail: koho-list[at]ml.omu.ac.jp
※上記の[at]は@に置き換えてください。








0 コメント:
コメントを投稿